使用eggnog-mapper注释基因组
参考资料:
github https://github.com/eggnogdb/eggnog-mapper
原始文献 https://doi.org/10.1093/molbev/msx148
一个中文教程 https://www.jianshu.com/p/9d38cb9f1d02
1. 实验室服务器下操作
1.1 默认参数运行
conda create -n eggnog
conda activate eggnog
conda install eggnog-mapper
mkdir /home/caigui/miniconda3/envs/eggnog/lib/python3.11/site-packages/data
download_eggnog_data.py # 一路y下去,解压后有48G数据库
# 运行,这里使用每个基因里最长的那个蛋白作为代表,并去除转录本序号以简化基因名称
nohup emapper.py -i xiaocuiyun_protein_represent_rename.fa -o xiaocuiyun_20230404_eggnog --tax_scope Viridiplantae --cpu 40 --dbmem &
# 好像不到十分钟就注释完了???
# 结果文件
less -S xiaocuiyun_20230404_eggnog.emapper.annotations
1.2 与interproscan结果进行比较
选一个基因SK03G20400,对比一下eggnog-mapper和interproscan的注释结果
################################################################################################################################################
# eggnog
SK03G20400 88036.EFJ33044 1.36e-67 222.0 28MZX@1|root,2QVBF@2759|Eukaryota,37RA9@33090|Viridiplantae,3GF59@35493|Streptophyta 33090|Viridiplantae K wuschel-related homeobox - GO:0000003,GO:0001067,GO:0003006,GO:0003674,GO:0003676,GO:0003677,GO:0003700,GO:0005488,GO:0006355,GO:0007275,GO:0008150,GO:0009653,GO:0009790,GO:0009791,GO:0009793,GO:0009888,GO:0009889,GO:0009933,GO:0009987,GO:0010014,GO:0010016,GO:0010072,GO:0010087,GO:0010154,GO:0010468,GO:0010556,GO:0019219,GO:0019222,GO:0022414,GO:0031323,GO:0031326,GO:0032501,GO:0032502,GO:0043565,GO:0044212,GO:0048316,GO:0048367,GO:0048507,GO:0048508,GO:0048532,GO:0048608,GO:0048731,GO:0048856,GO:0050789,GO:0050794,GO:0051171,GO:0051252,GO:0051301,GO:0060255,GO:0061458,GO:0065007,GO:0080090,GO:0090421,GO:0097159,GO:0140110,GO:1901363,GO:1903506,GO:2000112,GO:2001141 - ko:K18490 - - - - ko00000,ko03000 - - - Homeobox
# eggnog的go term
GO:0000003,GO:0001067,GO:0003006,GO:0003674,GO:0003676,GO:0003677,GO:0003700,GO:0005488,GO:0006355,GO:0007275,GO:0008150,GO:0009653,GO:0009790,GO:0009791,GO:0009793,GO:0009888,GO:0009889,GO:0009933,GO:0009987,GO:0010014,GO:0010016,GO:0010072,GO:0010087,GO:0010154,GO:0010468,GO:0010556,GO:0019219,GO:0019222,GO:0022414,GO:0031323,GO:0031326,GO:0032501,GO:0032502,GO:0043565,GO:0044212,GO:0048316,GO:0048367,GO:0048507,GO:0048508,GO:0048532,GO:0048608,GO:0048731,GO:0048856,GO:0050789,GO:0050794,GO:0051171,GO:0051252,GO:0051301,GO:0060255,GO:0061458,GO:0065007,GO:0080090,GO:0090421,GO:0097159,GO:0140110,GO:1901363,GO:1903506,GO:2000112,GO:2001141
################################################################################################################################################
################################################################################################################################################
# interproscan
SK03G20400 05c99823c90e86309c61782e8ea36ed9 323 CDD cd00086 homeodomain 102 164 1.04591E-11 T 07-01-2022 IPR001356 Homeobox domain GO:0003677
SK03G20400 05c99823c90e86309c61782e8ea36ed9 323 Gene3D G3DSA:1.10.10.60 - 101 168 3.1E-17 T 07-01-2022 - -
SK03G20400 05c99823c90e86309c61782e8ea36ed9 323 MobiDBLite mobidb-lite consensus disorder prediction 163 212 - T 07-01-2022 - -
SK03G20400 05c99823c90e86309c61782e8ea36ed9 323 ProSiteProfiles PS50071 'Homeobox' domain profile. 98 163 15.256396 T 07-01-2022 IPR001356 Homeobox domain GO:0003677
SK03G20400 05c99823c90e86309c61782e8ea36ed9 323 PANTHER PTHR46777 WUSCHEL-RELATED HOMEOBOX 13 36 217 9.1E-61 T 07-01-2022 IPR044559 WUSCHEL-related homeobox 13-like GO:0003700
SK03G20400 05c99823c90e86309c61782e8ea36ed9 323 Pfam PF00046 Homeodomain 102 162 3.7E-17 T 07-01-2022 IPR001356 Homeobox domain GO:0003677
SK03G20400 05c99823c90e86309c61782e8ea36ed9 323 SUPERFAMILY SSF46689 Homeodomain-like 94 166 4.28E-17 T 07-01-2022 IPR009057 Homeobox-like domain superfamily -
SK03G20400 05c99823c90e86309c61782e8ea36ed9 323 SMART SM00389 HOX_1 100 167 5.6E-13 T 07-01-2022 IPR001356 Homeobox domain GO:0003677
SK03G20400 05c99823c90e86309c61782e8ea36ed9 323 MobiDBLite mobidb-lite consensus disorder prediction 168 197 - T 07-01-2022 - -
SK03G20400 05c99823c90e86309c61782e8ea36ed9 323 Coils Coil Coil 60 80 - T 07-01-2022 - -
# interproscan的go term
GO:0003677,GO:0003700
################################################################################################################################################
怎么说呢,interproscan的go,eggnog那边都有。而且eggnog那边的注释非常简洁,就只有一句“wuschel-related homeobox”
1.3 与拟南芥同源基因注释比较
看一下这个基因的拟南芥同源基因在TAIR上的注释
# AtWOX13在org.At.tair.db的GO注释
"GO:0000976" "GO:0003700" "GO:0005634" "GO:0006355" "GO:0006631" "GO:0006996" "GO:0009888" "GO:0010025" "GO:0010087" "GO:0010629" "GO:0018193" "GO:0019748" "GO:0032787" "GO:0042546" "GO:0044255" "GO:0044550"
AtWOX13有以上16个go term,和eggnog的59个有4个交集。为什么eggnog可以自信地给出这么多GO注释?这就需要看eggnog的原始文献了。原始文献的摘要就说了,平均每个蛋白多41个term,确实。摘要还说它各个方面都比我之前用的interProScan好。
1.4 hmmer代替默认的dimand
用hmmer代替默认的dimand进行搜索试试。hmmer速度很慢,dimand速度很快,看看注释结果有什么区别。
# 用hmmer代替默认的dimand试一试
nohup emapper.py -i xiaocuiyun_protein_represent_rename.fa -o xiaocuiyun_20230404_eggnog --tax_scope Viridiplantae --cpu 40 --dbmem -m hmmer &
# 报错
# emapper.py: error: HMMER mode requires a target database (-d, --database).
nohup emapper.py -i xiaocuiyun_protein_represent_rename.fa -o xiaocuiyun_20230404_eggnog --tax_scope Viridiplantae --cpu 40 --dbmem -m hmmer -d euk &
# 还是报错
# 解决办法:需要下载数据库
# http://eggnog5.embl.de/#/app/downloads 查看taxID,下面的33090是Viridiplantae的ID
# 下载数据库
download_eggnog_data.py -H -d 33090
# 运行注释程序
nohup emapper.py -i xiaocuiyun_protein_represent_rename.fa -o xiaocuiyun_20230404_eggnog --tax_scope Viridiplantae --cpu 40 --dbmem -m hmmer -d 33090 &
################################################################################################################################################
SK03G20400 88036.EFJ33044 2.2e-85 280.0 28MZX@1|root,2QVBF@2759|Eukaryota,37RA9@33090|Viridiplantae,3GF59@35493|Streptophyta 33090|Viridiplantae K wuschel-related homeobox - GO:0000003,GO:0001067,GO:0003006,GO:0003674,GO:0003676,GO:0003677,GO:0003700,GO:0005488,GO:0006355,GO:0007275,GO:0008150,GO:0009653,GO:0009790,GO:0009791,GO:0009793,GO:0009888,GO:0009889,GO:0009933,GO:0009987,GO:0010014,GO:0010016,GO:0010072,GO:0010087,GO:0010154,GO:0010468,GO:0010556,GO:0019219,GO:0019222,GO:0022414,GO:0031323,GO:0031326,GO:0032501,GO:0032502,GO:0043565,GO:0044212,GO:0048316,GO:0048367,GO:0048507,GO:0048508,GO:0048532,GO:0048608,GO:0048731,GO:0048856,GO:0050789,GO:0050794,GO:0051171,GO:0051252,GO:0051301,GO:0060255,GO:0061458,GO:0065007,GO:0080090,GO:0090421,GO:0097159,GO:0140110,GO:1901363,GO:1903506,GO:2000112,GO:2001141 - ko:K18490 - - - - ko00000,ko03000 - - - Homeobox
################################################################################################################################################
# 将--tax_scope Viridiplantae改为--tax_scope 33090,试试有什么不同
nohup emapper.py -i xiaocuiyun_protein_represent_rename.fa -o xiaocuiyun_20230404_eggnog --tax_scope 33090 --cpu 40 --dbmem -m hmmer -d 33090 &
# 运行正常,估计和上面没什么不同
# 经过比对,结果确实一模一样
# 试试把-d也换成Viridiplantae
nohup emapper.py -i xiaocuiyun_protein_represent_rename.fa -o xiaocuiyun_20230404_eggnog --tax_scope Viridiplantae --cpu 40 --dbmem -m hmmer -d Viridiplantae &
# 报错,看来-d只能选33090,但是tax_scope可以选33090或者Viridiplantae
1.5 比较一下web版本的注释结果
# 试试官方的web版本的注释情况
# http://eggnog-mapper.embl.de/
# 显示注释完成了,但是结果文件下载全部失败,显示为空
# 过了几个小时再去看,发现文件又都有了,看来只是反应有点慢?
# 同一个基因的结果文件
################################################################################################################################################
SK03G20400 88036.EFJ33044 1.21e-68 224.0 28MZX@1|root,2QVBF@2759|Eukaryota,37RA9@33090|Viridiplantae,3GF59@35493|Streptophyta 35493|Streptophyta K wuschel-related homeobox - GO:0000003,GO:0001067,GO:0003006,GO:0003674,GO:0003676,GO:0003677,GO:0003700,GO:0005488,GO:0006355,GO:0007275,GO:0008150,GO:0009653,GO:0009790,GO:0009791,GO:0009793,GO:0009888,GO:0009889,GO:0009933,GO:0009987,GO:0010014,GO:0010016,GO:0010072,GO:0010087,GO:0010154,GO:0010468,GO:0010556,GO:0019219,GO:0019222,GO:0022414,GO:0031323,GO:0031326,GO:0032501,GO:0032502,GO:0043565,GO:0044212,GO:0048316,GO:0048367,GO:0048507,GO:0048508,GO:0048532,GO:0048608,GO:0048731,GO:0048856,GO:0050789,GO:0050794,GO:0051171,GO:0051252,GO:0051301,GO:0060255,GO:0061458,GO:0065007,GO:0080090,GO:0090421,GO:0097159,GO:0140110,GO:1901363,GO:1903506,GO:2000112,GO:2001141 - ko:K18490 - - - - ko00000,ko03000 - - - Homeobox
################################################################################################################################################
可以看到这里自动识别为了Streptophyta,而不是前面用的Viridiplantae。
看了看,上面那个查看taxID的链接内,也有Streptophyta的选项
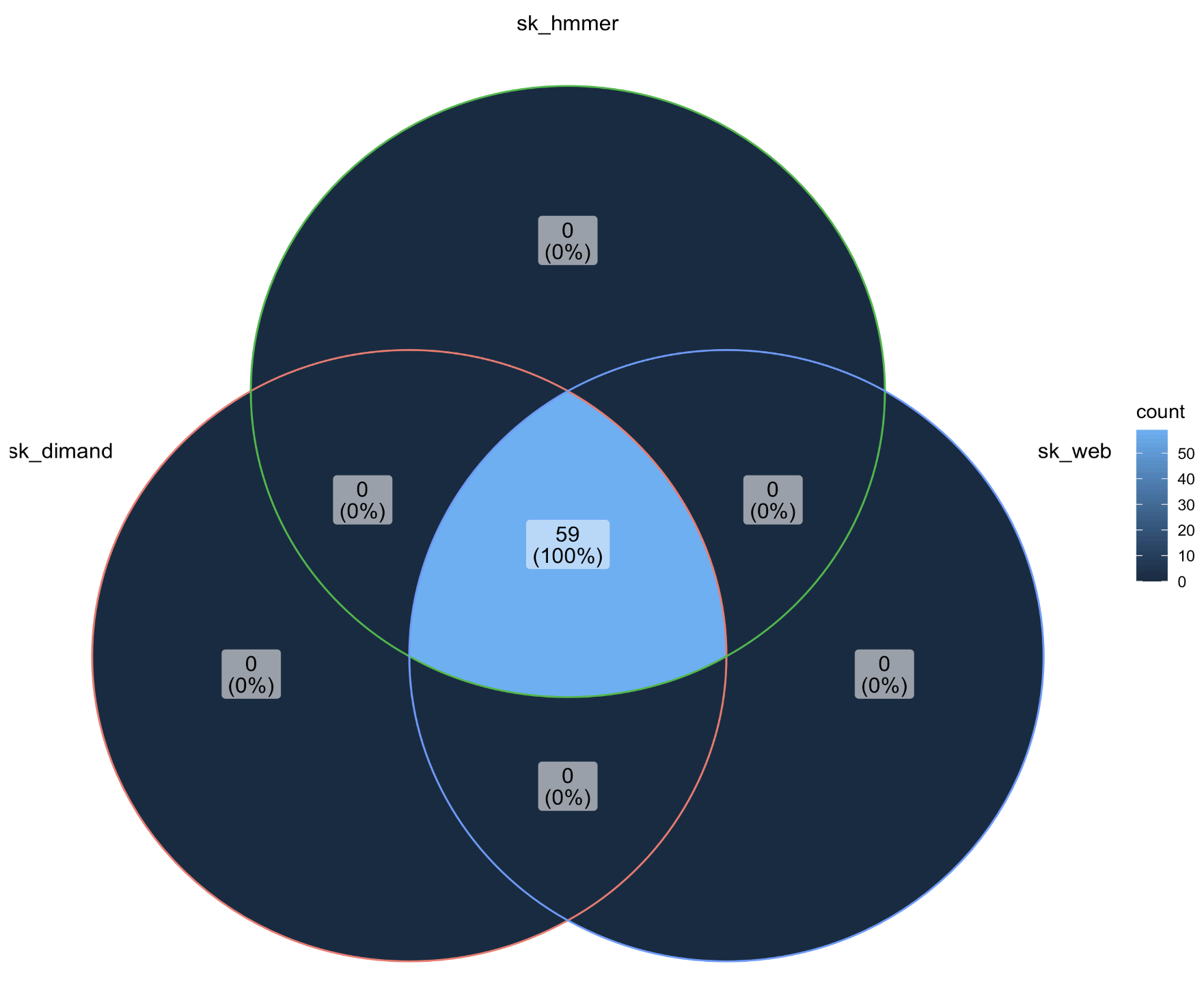
2. 最后画个维恩图看一下本地的dimand,本地的hmmer,和web版本默认参数下的注释区别
结果就是一模一样
3. 写一个小脚本将GO term提取出来
# 输出所有基因到一个文件
grep -o "SK[0-9Z]\{1,2\}G[0-9]\{5\}" xiaocuiyun_20230404_eggnog.emapper.annotations > sk.eggnog.id
# 跑个循环,找出每个基因的goterm,并按照"gene,go_term"的格式输出
for i in `cat sk.eggnog.id`
do
go_list=($(grep $i xiaocuiyun_20230404_eggnog.emapper.annotations | grep -o "GO:[0-9]\{7\}"))
for go in "${go_list[@]}"
do
echo "$i,$go" >> sk.eggnog.emapper.annot.GO.trem.20230616.csv
done
done
4. 总结
总的来说,用默认参数就可以了。
运行示例nohup emapper.py -i xiaocuiyun_protein_represent_rename.fa -o xiaocuiyun_20230404_eggnog --tax_scope Viridiplantae --cpu 40 --dbmem &
其中--tax_scope这个参数可以在这里查询
ps:--tax_scope这个参数好像不用也行哈